Mettre à jour Seatizen Atlas
Comment fonctionne Seatizen Atlas
Seatizen Atlas est une base de données SQLite stockée au format GeoPackage afin d'intégrer une dimension spatiale. Son but est de rassembler les métadonnées de chaque session Seatizen présente sur Zenodo.
Il est possible d'utiliser le logiciel DB Browser for SQLite pour explorer la base de données.
Voici son diagramme UML :
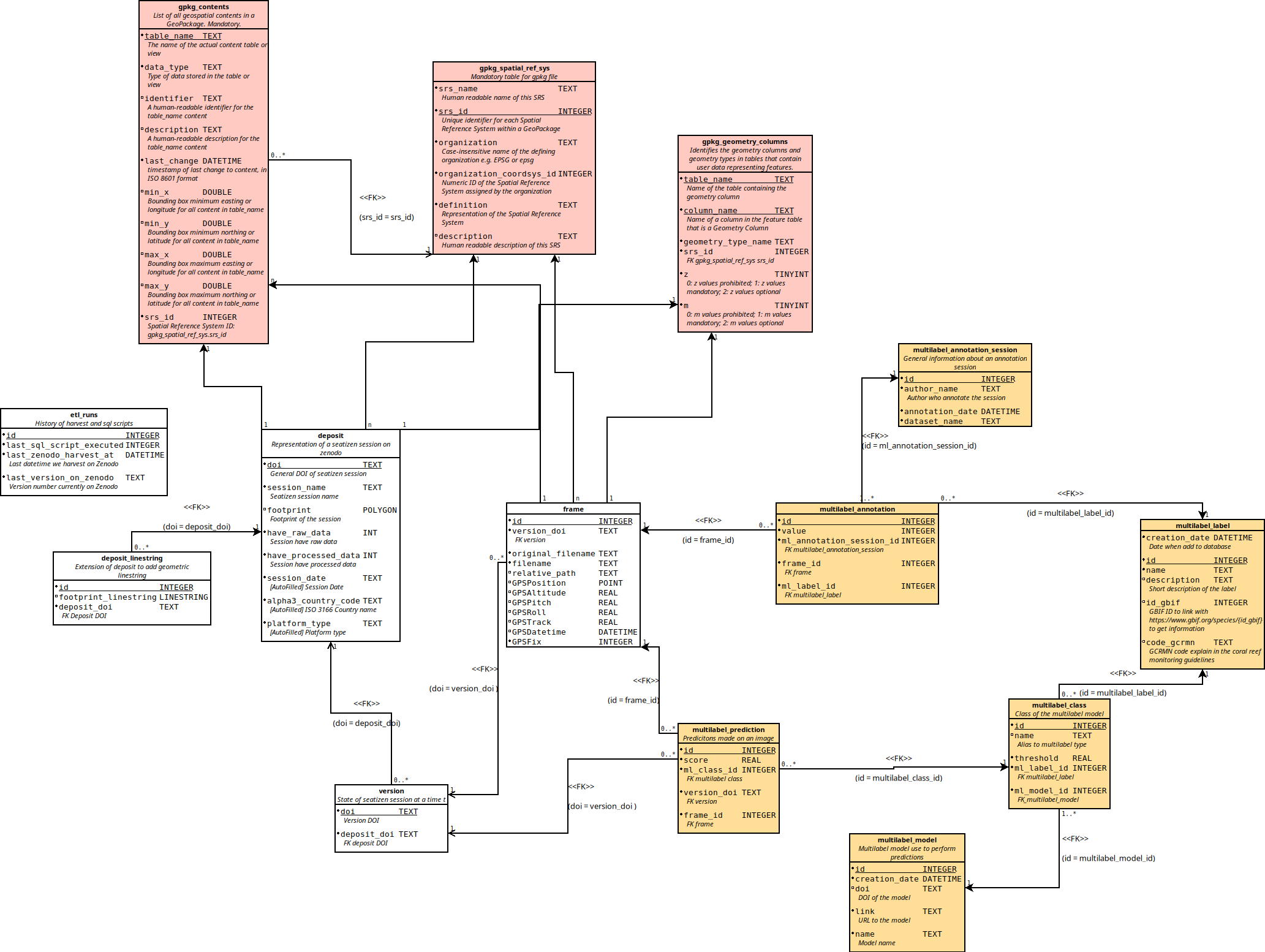
Elle stocke les métadonnées des images (table frame), ainsi que l'ensemble des métadonnées des prédictions et des annotations multilabels (partie en jaune).
Le script zenodo-manager.py permet de :
- Ajouter une ou plusieurs sessions dans la base de données. Attention : la session doit être présente sur le PC de l'utilisateur (sinon, voir
zenodo-auto-update.py). - Mettre à jour la base de données à partir d'un script SQL.
- Générer des fichiers CSV, Parquet et une archive au format Darwin Core.
- Téléverser la base de données sur Zenodo.
- Mettre à jour les métadonnées de la dernière version sur Zenodo.
- Ajouter des annotations (voir cheatsheet).
Le script zenodo-auto-update.py est plus simple d'utilisation. À partir d'une ou plusieurs communautés sur Zenodo, il met à jour la base de données locale en téléchargeant uniquement les sessions créées ou modifiées depuis le dernier moissonnage (voir table etl_runs du GeoPackage). Le script télécharge la dernière version des archives METADATA.zip et PROCESSED_DATA_IA.zip. Si les données de Zenodo ne sont pas encore présentes dans la base, il extrait les informations nécessaires puis supprime les dossiers locaux.
Cheatsheet de commandes
Exemples de commandes pour mettre en ligne ou à jour des sessions et la base de données.
Mettre les métadonnées à jour d'un dossier de sessions (-um) avec le fichier de métadonnées metadata/metadata.json
python zenodo-upload.py -efol -pfol /media/bioeos/D/202312_plancha_session -um -pmj metadata/metadata.json
Attention, la commande ci-dessus met à jour TOUTES les version d'un dépôt sur zenodo. Pour mettre à jour que la dernière version, il faut utiliser l'argument -umlv
Mettre en ligne un dossier de sessions. On aura une version de données brutes (-ur) et une version de processed_data (-up) avec les dossiers frames, metadata, et photogrammetrie. Le fichier de métadonnées à utiliser est ./metadata/metadata_recif3D.json. On force l'envoie des frames car on a pas utilisé Jacques pour filtrer les images inutiles (image sous-marine d'AUV).
python zenodo-upload.py -efol -pfol ./recif3D_sessions_folder/ -ur -up pmf -pmj ./metadata/metadata_recif3D.json --all_frames_without_filtering_by_useful
Récupère tous les deposits sur Zenodo et supprime ceux qui sont vierges, sans titre et en mode brouillon (Erreur lors d'un envoi, dépôt fantôme)
python zenodo-upload.py -eno -cd
Applique le script SQL numéro 3 sur le geopackage. Il doit être rangé dans le dossier https://github.com/SeatizenDOI/zenodo-tools/tree/master/src/sql_connector
python zenodo-manager.py -eno -ssn 3
Exporte tous les fichiers dérivés de la base de données (CSV, Parquet, darwin core, ...)
python zenodo-manager.py -eno -ulo
Régènère une base de données de zéro (-fr) localement (-ulo) et ajoute un dossier de sessions en forcant l'insertion des frames (n'utilise pas Jacques) (-ffi). Le programme ne génère pas les fichiers dérivés.
python zenodo-manager.py -efol -pfol /media/bioeos/E/2015_plancha_session/ -ulo -ffi -fr -ne # Force inserting frame in database with no export and regenerate database.
Envoie sur zenodo une nouvelle version de la base de donnée (-cp). Le programme ne regénère pas les fichiers dérivés.
python zenodo-manager.py -eno -ulo -cp -ne
Ajoute des annotations dans la base de données (-la).
python zenodo-manager.py -eno -ulo -la ../../Bioeos/annotations_some_image/Export_human/ -ne # Add frames and import annotations.
Un fichier suit un certaine nommage date_time__author-name__dataset-name.csv. Par exemple, voici le fichier 20240524_151409__victor-russias__annotations_over_predictions_1.csv:
FileName,Acropore_branched,Acropore_digitised,Acropore_sub_massive,Acropore_tabular,Algae_assembly,Algae_drawn_up,Algae_limestone,Algae_sodding,Atra/Leucospilota,Bleached_coral,Blurred,Dead_coral,Fish,Homo_sapiens,Human_object,Living_coral,Millepore,No_acropore_encrusting,No_acropore_foliaceous,No_acropore_massive,No_acropore_solitary,No_acropore_sub_massive,Rock,Rubble,Sand,Sea_cucumber,Sea_urchins,Sponge,Syringodium_isoetifolium,Thalassodendron_ciliatum,Useless
20230203_REU-GRANDFOND_ASV-1_00_1_197.jpeg,0,0,0,0,1,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,1,0,0,0,0
20230203_REU-GRANDFOND_ASV-1_00_1_201.jpeg,0,0,0,0,1,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,1,0,0,0,0
20230203_REU-GRANDFOND_ASV-1_00_1_212.jpeg,0,0,0,0,1,0,0,1,0,0,0,1,0,0,0,0,0,0,0,0,0,0,1,0,0,0,1,0,0,0,0
20230203_REU-GRANDFOND_ASV-1_00_1_218.jpeg,0,0,0,0,1,0,0,1,0,0,0,1,0,0,0,0,0,0,0,0,0,0,1,0,0,0,1,0,0,0,0
Si une image n'est pas présente déjà dans la table frames (lié à un DOI), on ne garde pas. Si un label d'annotation n'est pas présent dans la base de données, on n'ajoute pas l'annotation.